Introducción a R
¿Qué es R?
- R es un lenguaje de programación.
- R es un software de código abierto y libre.
- Cuando instalamos R, se instala su núcleo básico.
Sin embargo, gran parte de sus funciones adicionales se encuentran en lo que se conocen como “paquetes”
¿Por qué R?
Plataforma Versátil: R es compatible con una amplia gama de sistemas operativos, incluyendo Mac, Windows y Linux.
Lenguaje de Programación Completo: R es más que un simple software estadístico.
Promoción de la Investigación Reproducible: R no solo facilita la realización de análisis de datos, sino que también fomenta la investigación reproducible.
Comunidad Activa: R se mantiene actualizado gracias a su comunidad activa de usuarios y desarrolladores. Con cerca de 19950 paquetes disponibles en CRAN (Comprehensive R Archive Network).
Integración con Herramientas Bioinformáticas: R se integra de manera efectiva con herramientas bioinformáticas y formatos de datos procesados.
Capacidades Gráficas Sofisticadas: R ofrece capacidades gráficas avanzadas que te permiten crear visualizaciones atractivas y sofisticadas para representar tus datos de manera efectiva.
Popular en Estadísticas y Bioinformática: R es ampliamente reconocido y utilizado en la comunidad estadística, y su popularidad sigue creciendo en el ámbito de la bioinformática.
¿Cómo entender R?
- Hay una sesión de R corriendo. La consola de R es la interfaz entre R y nosotros.
- En la sesión hay objetos. Todo en R es un objeto: vectores, tablas, funciones, etc.
- Operamos aplicando funciones a los objetos y creando nuevos objetos.
La consola (o terminal)
Es una ventana que nos permite comunicarnos al motor de R. Esta ventana acepta comandos en el lenguaje de R y brinda una respuesta (resultado) a dichos comandos.
Por ejemplo, le podemos pedir a R que sume 1+1 así:
1+1La consola se distingue por tener el símbolo > seguido de un cursor parpadeante que espera a que le demos instrucciones (cuando recién abrimos R además muestra la versión que tenemos instalada y otra info).
Tu Consola debe verse más o menos así después del ejemplo anterior:
Scripts y el editor
Un script es un archivo de nuestros análisis que es:
- Un archivo de texto plano
- Permanente,
- Repetible,
- Anotado y
- Compartible
En otras palabras, un script es una recopilación por escrito de las instrucciones que queremos enviar a la consola, de modo que al tener esas instrucciones cualquiera pueda repetir el análisis tal cual se hizo.
Un script muy sencillo podría verse así:
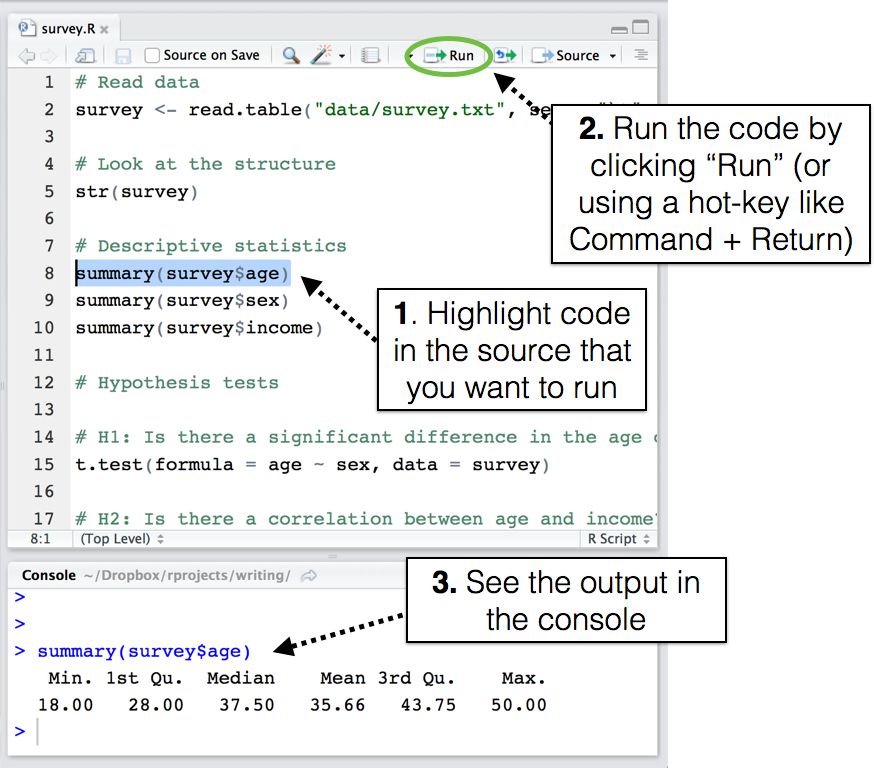
La idea es que en el editor de texto vayas escribiendo los comandos y comentarios de tu script hasta que haga exactamente lo que quieras.
Funciones Básicas de R
R Basico
Antes de adentrarnos en las funciones específicas para bioinformática, es fundamental comprender la sintaxis básica de R y los comandos esenciales que necesitas conocer. A continuación, se resumen estos conceptos clave:
Expresiones Matemáticas: Puedes realizar cálculos matemáticos simples, por ejemplo,
1+1.Strings de Texto: R permite trabajar con texto, como en el ejemplo
"¡Holaaaaa mundo!".Valores Lógicos: Puedes evaluar expresiones lógicas, por ejemplo,
1<5o2+2 == 5.Creación de Variables: Para almacenar datos, puedes crear variables u objetos, como en
x <- 5.Funciones: Las funciones son comandos que realizan tareas específicas en R.
Ejemplos de funciones comunes en R incluyen sum(), mean(), y log(). Para obtener ayuda detallada sobre la función log(), puedes utilizar el comando ?log.
Matrices
- Matrices
matrix(0, 3, 5) - Acceso a elementos e una matriz
[ , ]
Data frames
- Data frame
data.frame(x = c("a", "b", "c"), y = 1:3) - Acceso a elementos e una data.frame
[ , ],$
Cargar Archivos y Configurar WD
Cargar Archivos
Para cargar archivos de texto con filas y columnas en R, puedes utilizar la función read.table(). Cuando utilices read.table() u otras funciones similares, asume que tu Directorio de Trabajo (WD) es la ubicación donde reside tu script y utiliza rutas relativas para acceder a los archivos que deseas cargar.
Configurar el Directorio de Trabajo (WD)
Es una buena práctica que tu Directorio de Trabajo sea la ubicación donde reside el script en el que estás trabajando. Para determinar cuál es tu Directorio de Trabajo actual en R, puedes utilizar la función getwd().
Si necesitas cambiar manualmente tu Directorio de Trabajo, utiliza la función setwd(). Sin embargo, ten en cuenta que debes realizar esta acción en la Consola de R, no en tu script.
Una trampa útil en RStudio para establecer automáticamente tu Directorio de Trabajo en la ubicación de tu script es seleccionar la opción: Session > Set Working Directory > To source file location
Trabajar con Paquetes y Datos
Puedes explorar una amplia gama de paquetes en el repositorio CRAN (Comprehensive R Archive Network) en CRAN. Algunos paquetes están diseñados específicamente para la bioinformática, como adegenet y ape. También puedes encontrar una lista de paquetes relacionados con la genética estadística en CRAN Task Statistical Genetics.
Para instalar un paquete en tu máquina, utiliza el comando install.packages.
Una vez que el paquete está instalado, debes cargarlo en la sesión de R utilizando la función library cada vez que necesites utilizarlo en un análisis específico.
Crear Funciones Propias
La capacidad de crear tus propias funciones en R es esencial para modularizar tu análisis y mantener tu código organizado. Aquí tienes una guía básica para crear y utilizar funciones propias:
Uso de source():
- La función
source()te permite ejecutar un script de R dentro de otro script de R. Esto es útil para modularizar tu código y reutilizar funciones personalizadas. Puedes llamar a un script externo utilizandosource()de la siguiente manera:
source("nombre_del_script.r")Asegúrate de que tu Directorio de Trabajo getwd() sea la ubicación correcta para el archivo que deseas cargar.
Crear una Función Propia:
- Para crear tu propia función en R, sigue el siguiente esqueleto básico:
nombre_de_tu_funcion <- function(arg1, arg2, ...) {
# Declaraciones y operaciones
return(resultado)
}nombre_de_tu_funcion: Es el nombre que asignas a tu función.arg1, arg2, ...: Representan los argumentos que tu función acepta.# Declaraciones y operaciones: Aquí defines las operaciones que deseas realizar dentro de la función.return(resultado): Utilizareturn()al final de la función si deseas que la función devuelva un resultado. De lo contrario, la función se ejecutará pero no devolverá ningún valor.
Ejemplo de Función Propia:
- Aquí tienes un ejemplo de una función propia que hace un conteo de los datos dentro de un array:
conteo_datos <- function(array) {
x <- table(array)
return(x)
}Al modularizar tu código utilizando funciones propias y source(), puedes mejorar la organización de tu análisis, reutilizar código y compartir funciones útiles con otros usuarios.
Manipulación y Limpieza de Datos
La manipulación y limpieza de datos son pasos fundamentales antes de realizar análisis en R. A continuación, exploraremos algunas de las funciones más comunes de dplyr junto con ejemplos de uso:
Instalación y Carga de dplyr
Antes de comenzar, asegúrate de tener instalado el paquete dplyr. Si aún no lo has hecho, puedes instalarlo y cargarlo de la siguiente manera:
install.packages("dplyr")
#install.packages("tidyverse")
library(dplyr)Filtrar Filas
La función filter() se utiliza para seleccionar filas específicas de un conjunto de datos en función de condiciones específicas.
Ejemplo:
# Filtrar datos para seleccionar solo las filas donde la columna "edad" es mayor o igual a 18.
nuevos_datos <- datos %>% filter(edad >= 18)Seleccionar Columnas
Con select(), puedes elegir las columnas que deseas incluir en tu conjunto de datos y descartar las que no necesitas.
Ejemplo:
# Seleccionar solo las columnas "nombre" y "edad" del conjunto de datos.
datos_seleccionados <- datos %>% select(nombre, edad)Crear Nuevas Columnas
La función mutate() te permite crear nuevas columnas en tus datos basadas en cálculos o transformaciones de las columnas existentes.
Ejemplo:
# Crear una nueva columna "edad_doble" que contenga el doble de la edad.
datos_con_nueva_columna <- datos %>% mutate(edad_doble = edad * 2)Agregar y Resumir Datos
Para realizar agregaciones y resúmenes de datos, group_by() se utiliza para agrupar datos en función de una o más columnas, y summarize() permite calcular estadísticas resumidas para cada grupo.
Ejemplo:
# Calcular la media de la edad para cada género.
resumen_edad_por_genero <- datos %>%
group_by(genero) %>%
summarize(media_edad = mean(edad))Ordenar Filas
La función arrange() se utiliza para ordenar las filas de un conjunto de datos en función de una o más columnas.
Ejemplo:
# Ordenar el conjunto de datos por la columna "edad" de forma ascendente.
datos_ordenados <- datos %>% arrange(edad)Filtrar Filas Únicas
Si deseas eliminar filas duplicadas y quedarte solo con filas únicas, distinct() es la función adecuada.
Ejemplo:
# Eliminar filas duplicadas basadas en la columna "nombre".
datos_unicos <- datos %>% distinct(nombre)Creación de Gráficos en R
ggplot2 es una poderosa librería de R para crear gráficos de alta calidad. Aquí exploraremos algunas de las funciones clave y cómo utilizarlas para generar visualizaciones efectivas.
Instalación y Carga de ggplot2
Antes de comenzar, asegúrate de tener instalado el paquete ggplot2. Si aún no lo has hecho, puedes instalarlo y cargarlo de la siguiente manera:
install.packages("ggplot2")
library(ggplot2)Para crear un gráfico básico con ggplot2, debes especificar un conjunto de datos y mapear las variables a los estéticos (como x e y) utilizando la función ggplot(). Luego, puedes agregar capas de geometrías para representar los datos.
# Crear un gráfico de dispersión simple.
ggplot(data = datos, aes(x = edad, y = puntaje)) +
geom_point()Personalización de Gráficos
ggplot2 permite una amplia personalización de los gráficos. Puedes ajustar colores, etiquetas, escalas y más.
Ejemplo:
# Personalizar el gráfico de dispersión.
ggplot(data = datos, aes(x = edad, y = puntaje)) +
geom_point(color = "blue", size = 3) +
labs(title = "Relación entre Edad y Puntaje",
x = "Edad",
y = "Puntaje")ggplot2 es versátil y puede crear varios tipos de gráficos, como histogramas, barras, líneas y más. Solo debes ajustar la función geom_ según el tipo de gráfico deseado.
Histograma
# Crear un histograma de la variable "edades".
ggplot(data = datos, aes(x = edad)) +
geom_histogram()Gráfico de Barras
# Crear un gráfico de barras para contar la frecuencia de géneros.
ggplot(data = datos, aes(x = genero)) +
geom_bar()Gráfico de Líneas
# Crear un gráfico de líneas para seguir cambios a lo largo del tiempo.
ggplot(data = datos, aes(x = tiempo, y = valor)) +
geom_line()Facetas
Las facetas te permiten dividir un gráfico en múltiples paneles según una variable. Esto es útil para explorar datos segmentados.
Ejemplo:
# Crear un gráfico de dispersión facetado por género.
ggplot(data = datos, aes(x = edad, y = puntaje)) +
geom_point() +
facet_wrap(~genero)Cómo Citar en R
Si necesitas citar R o un paquete en particular en tu trabajo, puedes utilizar los siguientes comandos:
Citar R:
citation("base")Citar un paquete en particular:
citation("NombrePaquete")